生命情報システム科学分野とは?
私たちの研究室では、ゲノムの配列情報に代表される「生命情報」を情報科学的に扱い、生命現象の理解を目指しています。
具体的には、ゲノム配列の解析、遺伝子の発現量解析、タンパク質の機能解析などです。
従来の生物学は個別の議論を蓄積し、どんどん教科書を分厚くする枚挙の学問でした。もちろん個別論を見る楽しさは生物学の醍醐味ですが、生命情報ビックバンとも言える急激な生命情報の増加により、生物学は変わりつつあります。
例としてヒトゲノム解析の場合を見てみます。
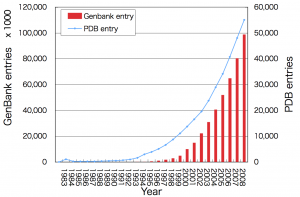
2001年にヒトゲノムの解析がほぼ終了しましたが、この解析には11年の歳月と4億3700万ドル(約445億円)の費用がかかりました。到底1つの研究室でできる規模ではありません。しかし、2007年にはCraig Venter個人のゲノムが100億ドル・4年間で解析され、2008年にはたった100万ドル・1年でJames Watsonのゲノムの解読が行われ、今後数年で、1000ドル(約10万円)で個人のゲノムが解析されるようになると考えられています。これぐらいの額になると、1つの研究室で、数十人のゲノムを解析することも可能になってきます。これは、これまで平均値(多くの人のゲノムの集まり)の情報しか無かったのに、個別のケースのデータが観察できる状態になるので、劇的に状況は変わります。
データが増えることは、冗長なデータでなければ基本的に良いことですが、データが増えれば増えるほどその処理は大変になってきます。特に、せっかく情報が増えてもちゃんと処理しないと貴重な情報を失いかねません。
例として最近我々が行った研究を簡単に紹介します。
遺伝子が同時に発現しているかどうかを考える際に「共発現」という重要な概念があります。これは、それぞれの遺伝子の発現量データから計算される物ですが、従来は単純に遺伝子ペアの発現量の相関係数を用いて共発現を定量化していました。しかし、相関係数には様々な問題があります。一番大きな問題は、相関係数は少数のハズレ値の影響が大きいことです。そこで我々は、最初に主成分解析を行って発現量を主成分空間に射影し、主な主成分を順に抜くことで、小数のデータの影響を除いた多次元共発現度指標を考案し、この指標が従来指標に比べて遺伝子の機能的関連を上手く表現していることを示しました(Kinoshita & Obayashi, 2009, Bioinformaticsと下図参照)。
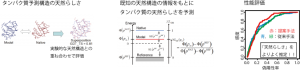
タンパク質立体構造予測における天然らしさの評価
遺伝子の分子としての実体はタンパク質であり,それぞれが3次元の「かたち」を持っています.これをタンパク質の天然構造といいます.タンパク質のアミノ酸配列から,その天然構造を予測することは生命情報学の大きな課題です.そして,この課題を解決するためには様々な予測構造の「天然らしさ」をきちんと評価しなければなりません.最近我々は,今までに知られているタンパク質の天然構造についての知見をもとに,新しいタンパク質の立体構造がどのくらい天然らしいかを評価する方法を開発しました.そしてこの方法が,機械学習などを用いた既存の方法と比較してよりよく「天然らしさ」を表現していることを確認しました(Shirota et al. 2011, Proteins).
このように、我々の研究室では生命情報ビックバンの時代における新しい生物学を目指して、生命情報を上手に解析することで、生命システムの理解を目指しています。
遺伝子及び遺伝子産物であるタンパク質の機能推定に関する専門的な説明は、英語ですがこちらをご覧下さい。