情報学的にどのような課題があるか?
生命情報ビッグバンの時代に、情報学的に生命現象を理解するために何が求められているのでしょうか?
大きな柱は3つあると思います。
- 大量データの処理
とにかく大量のデータをどのように扱うかがまず問題になります。例えば、ヒトの遺伝子が約3万個。遺伝子の共発現を考えたときはそのすべての組み合わせが問題になるので、9×10**8ペアを扱う必要があります。また、共発現を計算するための発現量データは、数千から数万実験のデータを利用するので、(遺伝子の数)x(実験数)だけの発現量データがあります。データは、英国にあるArrayExpressなどに納められていますから、そこから必要なデータをネットワークを介して取得し、色々な処理をして、解析を行い、最終的にはデータベース化して公開する事になります。ほぼ同規模のデータを、数種類の生物種に対して適応する事を考えると、扱うデータ数の大きさはだいたい想像できると思います。これらの大量データをディスク上にどのように配置するのかとか、どのように効率よくDBMSに納めるかなど、表には出ない部分で技術的に考えるべき課題は多いですが、これらをすべて乗り越えた暁に、生物学的におもしろい現象が見えてきます。 - データの可視化の必要性
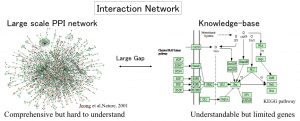
データが多くなると、全体が見えにくくなります。これらをすまく可視化することで、全体の特徴を捉えることが出来るときがあります。これがうまくいくと、非常に直感的に生物学的現象に迫る知見を得ることができることがあります。例えば、遺伝子の相互作用ネットワークをどのように書くか(生物学的に意味のあるノードとエッジを残して、適切に配置する問題)は、現在直面している大きな課題です。 - 大量データに対する検索
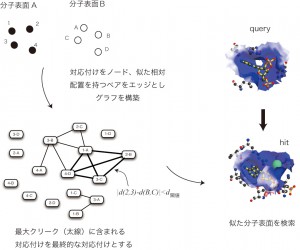
データが多くなると、いかに早く検索するかが重要な課題になってきます。特に生物学では「似ている遺伝子は機能的にも似ている」事が多いので、似ている遺伝子を探すというのは重要な課題です。配列の類似性検索でもさらなる高速化が必要ですし、遺伝子産物であるタンパク質では、その立体構造似ているかどうかが非常に重要なことがあるのですが、タンパク質の立体構造(=3次元オブジェクト)の類似性検索はまだまだ研究の余地が数多く残されています。我々が開発を行った(Kinoshita & Nakamura, Protein Science, 2003)、グラフ理論を応用した分子表面構造の類似性検索法もまだまだ高速化の余地があります。 - 実験を行う研究者が使えるwebデータベースの構築
生物学のみならずすべての自然科学では、すべての学説は実験で検証される必要があります。そのため、解析を行った結果は、実際に実験の研究者が利用できる、使い勝手の良いデータベースとして公開されることが非常に大切です。我々の研究室でも数多くのデータベースを構築してきましたが、今後も、より実験家の役にたつデータベースの構築を行い、生物学の理解を進めることを目指しています。我々が開発した主なデータベース or Web services